
VIRALERT project
COntexte
La fréquence et l’ampleur des épidémies d’origine virale ont augmenté au cours des dernières décennies. Une inter-connectivité mondiale forte facilite indéniablement la propagation des maladies infectieuses à travers les continents. Les changements globaux affectent également la répartition géographique des vecteurs de maladies ainsi que les interfaces entre les humains et les animaux, offrant ainsi davantage d’opportunités aux agents pathogènes de franchir la barrière entre espèces.
Les infections virales – telles que celle due au SARS-CoV-2 – provoquent fréquemment une large gamme de symptômes, et les formes graves de la maladie ne constituent souvent que la partie émergée de l’iceberg. Cela peut entraîner un délai entre l’émergence des agents pathogènes et leur détection par les systèmes classiques de surveillance des maladies infectieuses, qui reposent sur les signes cliniques.
L’émergence et la propagation du SARS-CoV-2 ont illustré les défis actuels en matière de surveillance, de gestion et de contrôle des maladies infectieuses, mais elles ont également favorisé des innovations. Si la surveillance des virus dans les eaux usées était déjà utilisée dans le cadre des stratégies d’éradication du virus de la polio, la pandémie COVID-19 a permis de mettre davantage en évidence son intérêt en tant qu’outil rentable et non invasif pour fournir une image de la circulation des agents pathogènes à l’échelle d’une population entière.
Toutefois, dans un contexte « Une Seule Santé » reconnaissant l’interdépendance de la santé humaine, animale et environnementale, la surveillance devrait aller encore plus loin. Les eaux usées reflètent principalement le microbiome humain, mais excluent les populations animales. L’épidémiologie des eaux de surface, exploitant la contamination fécale environnementale comme marqueur de la santé des animaux sauvages et domestiques, a été rarement explorée pour combler cette lacune.
objectifs
S’appuyant sur la longue expertise du groupe de Virologie Clinique et Appliquée du LIH en matière de surveillance des maladies humaines et animales, ainsi que sur les collaborations antérieures entre le LIH et le LIST dans le domaine de la surveillance environnementale des maladies infectieuses (cf. projets financés par le FNR « SENSORLUX » et « CORONASTEP+ »), le projet VIRALERT a proposé une approche intégrative innovante visant à ouvrir la voie à la surveillance de la circulation de virus à fort potentiel épidémique et pandémique, de manière non invasive et rentable.
À cette fin, le projet VIRALERT visait à :
- surveiller une gamme élargie de virus dans les eaux usées, incluant des virus entériques et respiratoires ;
- surveiller la santé des populations dans un contexte post-pandémique, dans lequel la circulation d’autres virus a été affectée par les mesures de contrôle de la pandémie ;
- couvrir la santé des animaux sauvages et domestiques en étudiant les populations virales dans les eaux douces ;
- explorer le potentiel de différentes technologies présentant des caractéristiques de sensibilité et de spécificité variables pour la détection moléculaire et le séquençage des virus de la grippe, du virus respiratoire syncytial humain et bovin, des coronavirus saisonniers humains et animaux, des entérovirus, des norovirus et des flavivirus transmis par les moustiques ;
- tirer parti des technologies de séquençage de nouvelle génération (séquençage à haut débit) afin d’explorer la possibilité de détecter un éventail plus large de virus.
À long terme, le renforcement de la surveillance environnementale fournira un outil supplémentaire aux autorités de santé publique et de santé animale pour compléter les systèmes de surveillance existants et contribuer à la réduction de la charge des maladies infectieuses.
CONTACT
Hübschen
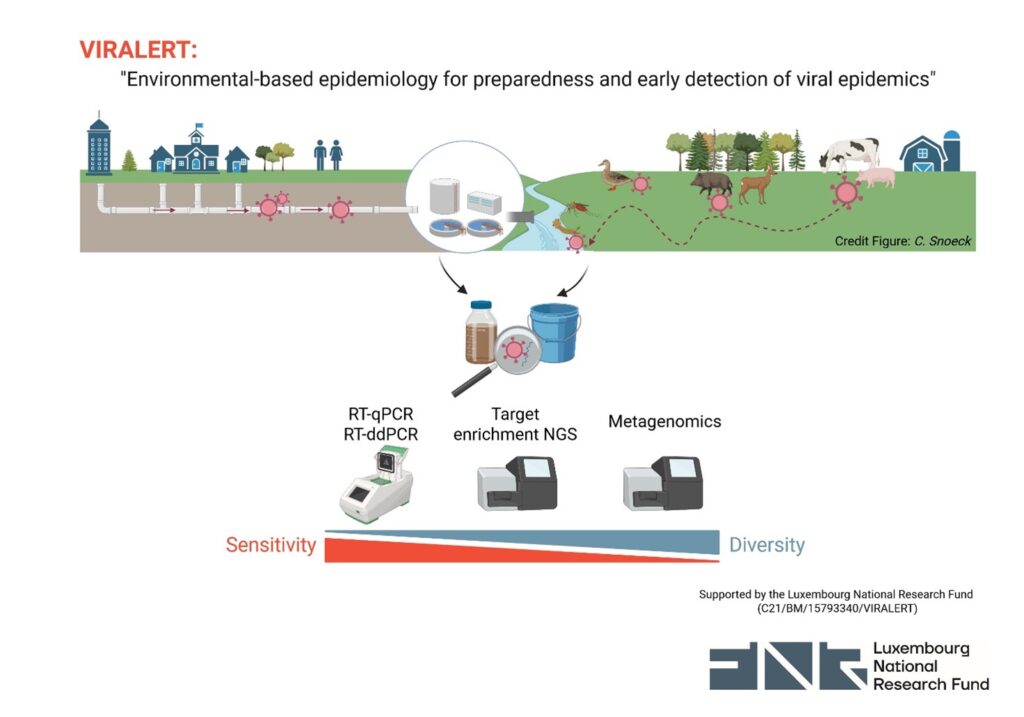
Vue schématique du cadre général du projet VIRALERT et des technologies employées (réalisée avec BioRender).
Abréviations : RT-qPCR, réaction de polymérase en chaîne en temps réel après transcription inverse; RT-ddPCR, PCR digitale en gouttelettes après transcription inverse; NGS, séquençage à haut débit.
Partenaires du projet



Actualités associées

Supported by the Luxembourg National Research Fund (C21/BM/15793340/VIRALERT)
